
联系我们
地址: 安徽省合肥市高新区望江西路5089号
邮编: 230026
电话: 0551-62391679
邮箱: iai@iai.ustc.edu.cn
National Science Review | 合肥综合性国家科学中心人工智能研究院/中国科大/安徽大学 开辟TB级小鼠全脑神经元活动印迹成像数据分析新方法
文章来源:本站原创 发布时间:2024-06-14 15:43:38 文章作者:本站编辑
全脑神经元活动印迹图谱分析是鉴定特定刺激激活神经元在全脑分布的主要途径,是开展神经环路功能研究的前提。随着组织透明化、光片显微成像技术的快速发展,研究人员能够快速批量化采集小鼠全脑神经细胞活动数据,但从若干TB全脑显微成像数据中系统分析神经活动印迹存在诸多挑战。
2024年6月12日,National Science Review正式发表了由中国科学技术大学/合肥综合性国家科学中心人工智能研究院王浩副研究员、吴枫教授、毕国强教授与安徽大学汤进教授合作完成的“NEATmap: a high-efficiency deep learning approach for whole mouse brain neuronal activity trace mapping”工作,报道了一种用于小鼠全脑神经元活动印迹图谱分析的高效深度学习方法,开发的NEATmap(NEuronal Activity Trace mapping)软件,可在约6个小时内自动化完成一个小鼠全脑的c-Fos神经元分析。结合作者团队前期发展的VISoR高速三维全脑显微成像技术与NEATmap分析方法,作者们高效、批量化成像分析了多组多只动物差异化的全脑神经元激活。
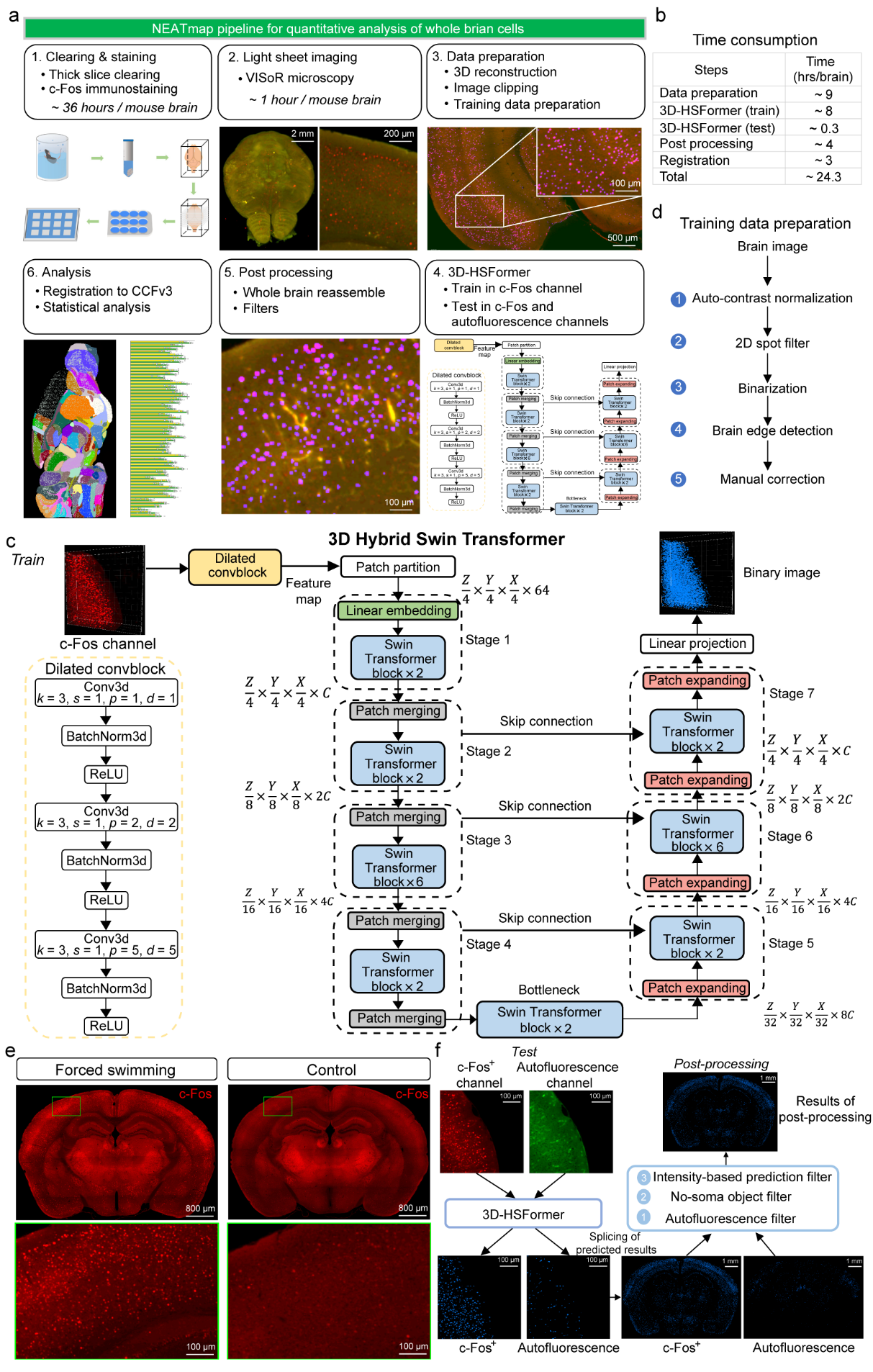
图1. NEATmap:组织透明化、免疫荧光、三维全脑成像、细胞分割和定量分析
研究人员所提出的方法主要包括三个主要步骤:首先,对小鼠脑进行组织透明化、免疫荧光染色和VISoR高速三维全脑显微成像,高效实现c-Fos信号和组织自发荧光的高分辨双通道成像。其次,利用搭建的3D Hybrid Swin Transformer(3D-HSFormer)深度学习模型,在约20分钟内完成全脑范围内激活神经元的自动化分割,并综合自发荧光通道分割结果以及细胞形态等信息剔除误分割的c-Fos+细胞,实现高精度的c-Fos+细胞分割。最后,将全脑图像数据和分割结果配准到Allen标准脑图谱(CCFv3)上,对在不同级别的脑区中的神经元活动印迹、动物个体差异以及左右半球的神经元激活情况进行全面分析。
在细胞分割的效率和精确度方面,与先进的基线模型和生物图像分析工具进行对比, NEATmap不仅显著提高了分割效率,也取得了更高的分割精度。
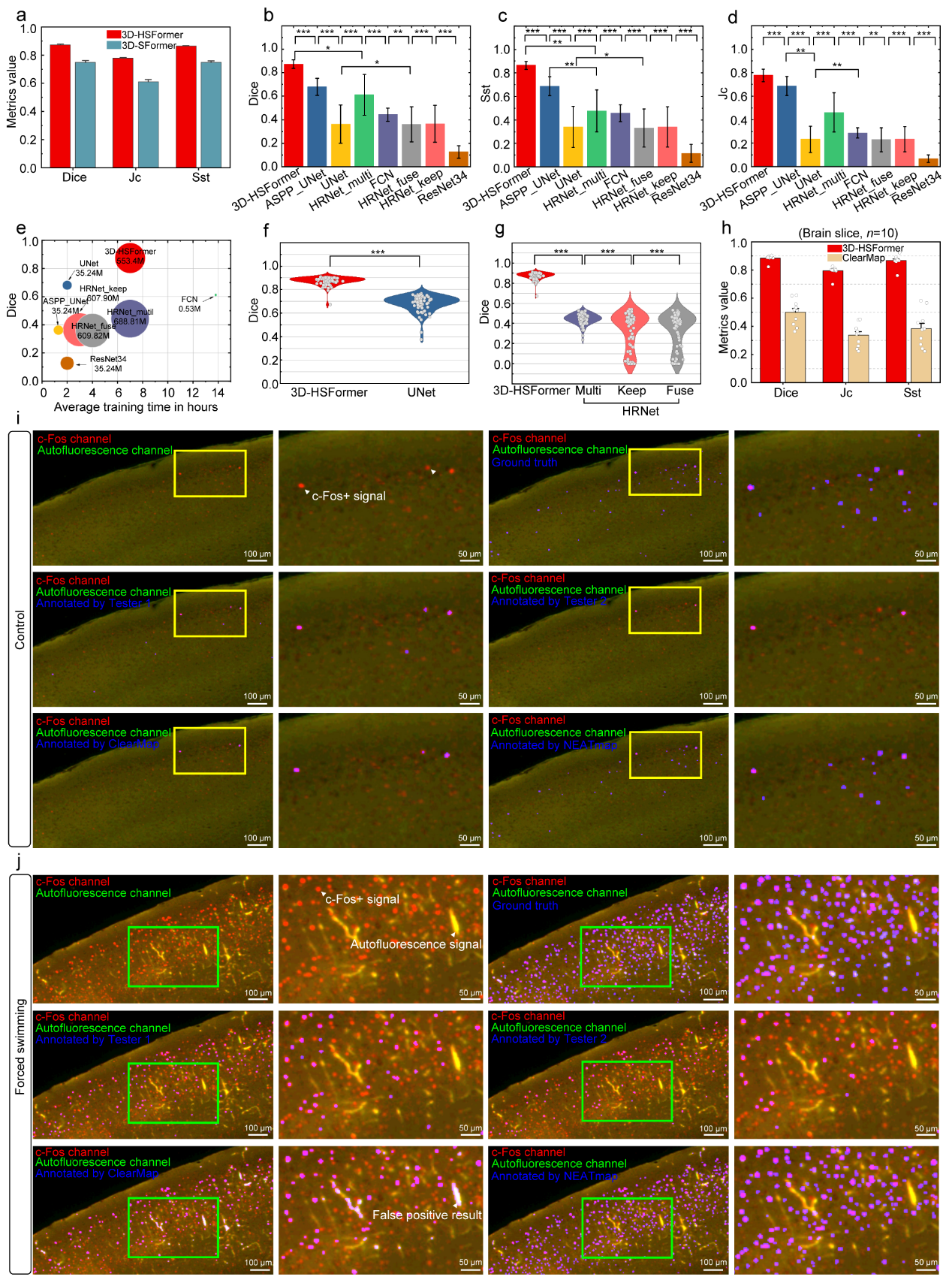
图2. NEATmap性能评估以及与其他方法的比较
在应用方面,研究人员将NEATmap应用到两种动物行为学模型:强迫游泳测试和急性社交失败情绪压力测试,分别进行了系统性的全脑神经元活动分析。例如在社交失败情绪压力测试中,研究人员发现社交失败组相比情绪压力组在丘脑和下丘脑的多个亚区表现较高的个体差异性。
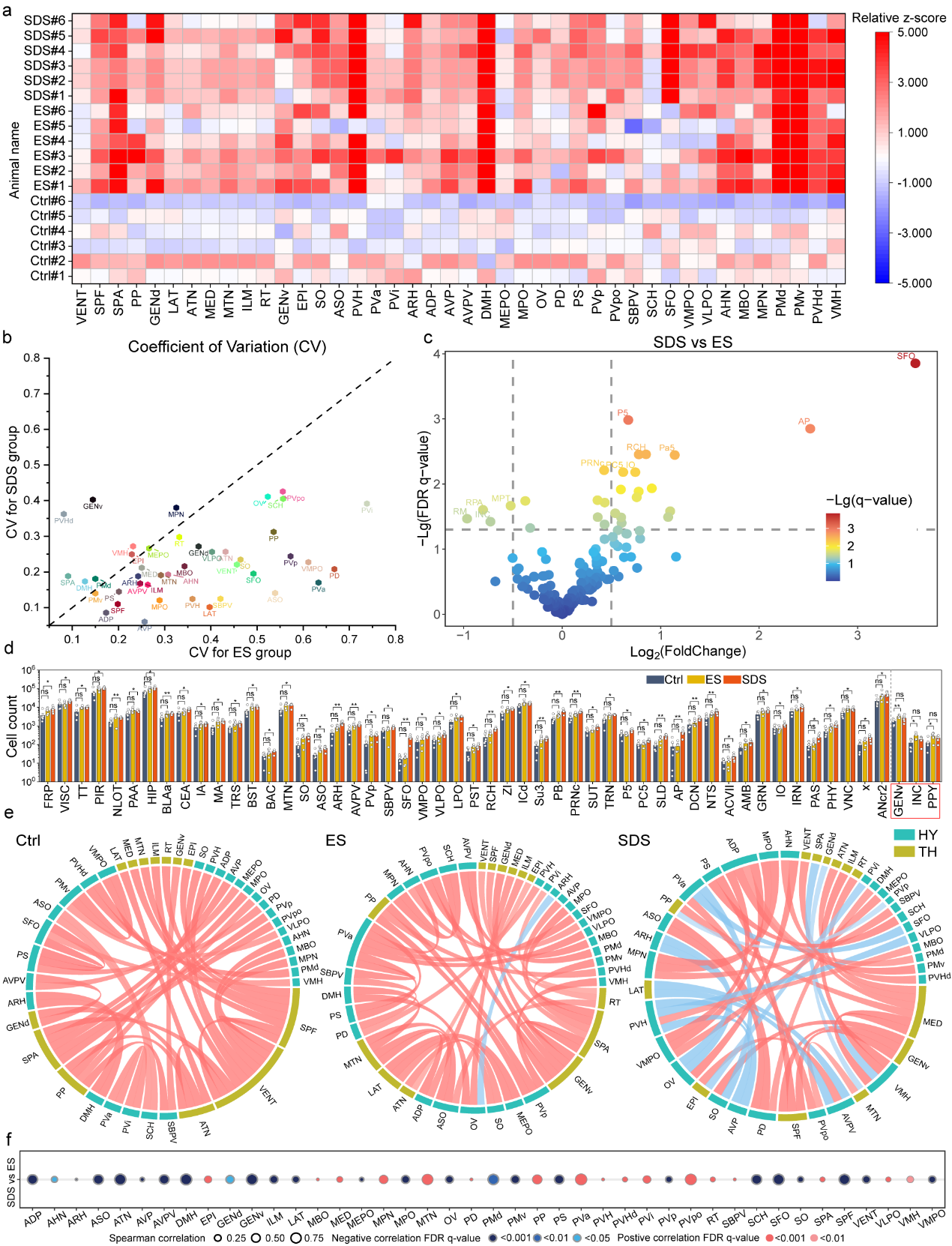
图3. 丘脑和下丘脑区域神经元激活的动物个体变异性和相关性分析。
为了促进神经科学领域对全脑神经元活动印迹的分割和系统分析,作者团队开发了用户友好性的GUI软件,提供了用户使用指南和教学视频以方便神经科学研究者使用。链接:https://github.com/mesobrain/NEATmap_software。
合肥综合性国家科学中心人工智能研究院/安徽大学联合培养博士生郑伟杰同学与中国科大副研究员母华伟博士为论文共同第一作者;安徽大学/合肥综合性国家科学中心人工智能研究院汤进教授、中国科学技术大学/合肥综合性国家科学中心人工智能研究院的毕国强教授、吴枫教授、王浩副研究员为论文共同通讯作者。
文章链接:https://academic.oup.com/nsr/article/11/5/nwae109/763569
文稿来源:脑成像平台





